Geist und Materie
Wissenschaftliche Daten (in unserem Falle z.B. EEG, fMRI oder Verhaltensdaten) enthalten Muster, die wir meist mit bloßem Auge nicht sehen können. Ausgefeilte Analysemethoden erlauben jedoch, solche Muster zu identifizieren, ausgefeilte grafische Methoden ihre Darstellung. Auswertungsergebnisse hängen dabei immer sehr stark von der gewählten Methode, ihren technischen Einschränkungen und von den Methoden-spezifischen Annahmen ab. Methoden der künstlichen Intelligenz (KI) – und speziell des maschinellen Lernens (ML) – zeigen eine zunehmende Leistungsfähigkeit bei der Analyse von wissenschaftlichen Daten. ML-Methoden werden mittlerweile in neurowissenschaftlicher und dort speziell in der klinischen Forschung, z.B. bei der Segmentierung von 3D Tomographiebildern, oder bei Klassifikationsaufgaben in der Psychopathologie und anderen Bereichen verwendet (Milletari et al. 2016, Qureshi et al. 2019, Rad & Furlanello 2016).
Allerdings geht die oft höhere Leistungsfähigkeit von ML-Lösungen gegenüber früheren Standartmethoden mit einem langwierigen Training des künstlichen Netzwerks einher. Dabei gewinnen leistungsfähige Computer und insbesondere Hochleistungs-Grafikkarten immer mehr an Bedeutung. Im Herbst 2019 konnten wir in diesem Zusammenhang erfolgreich ein Eucor–Seeding Projekt zur Auswertung klinischer Daten einwerben. Darüber hinaus bekamen wir im Rahmen einer Ausschreibung der Firma NVIDIA den Zuschlag für eine Hochleistungs-Grafikkarte (Nvidia Titan V). Unter Einsatz dieser Grafikkarte werten wir aktuell vorhandene EEG-Daten mit KI-Methoden aus und entwickeln künstliche neuronale Netze zur Analyse der Quellen, welche dem EEG zugrunde liegen. Unter Einsatz dieser Grafikkarte werten wir aktuell vorhandene EEG-Daten mit KI-Methoden aus (Abb. 1). So konnten wir unter Einsatz von KI eine Methode zur Quellenlokalisation von EEG-Signalen entwickeln (Hecker et al., 2021, 2023), aber auch EEG-Daten mit Hilfe von KI-Methoden genauer analysieren (z. B. Wilson et al., 2023).
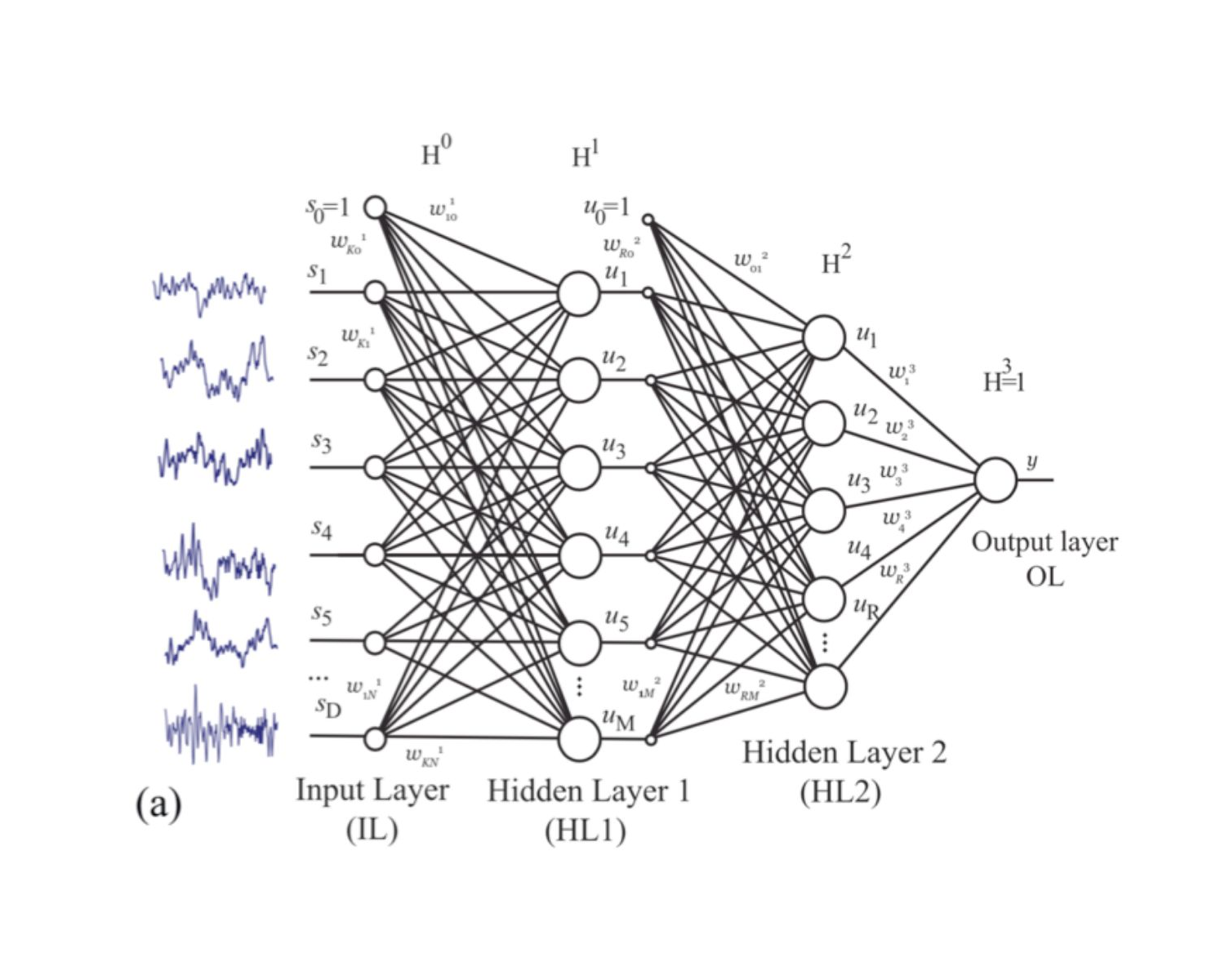
Abb. 1
Beispiel für ein Feedforward / Fully Connected Neural Network (aus Hramov et al. 2019). Blaue Kurven (links) sind Beispiele einzelner EEG Kurven von einer Versuchsperson. si, ui und y sind Neurone eines künstlichen Netzwerks. ωi symbolisieren Gewichtungen für die Informationsübertragung zwischen Neuronen aufeinander folgender Ebenen. Während des Trainings eines solchen künstlichen Netzwerks werden die ωi schrittweise verändert um die Netzwerkleistung zu verbessern.
Publikationen
Hecker, L., Tebartz Van Elst, L., & Kornmeier, J. (2023). Source localization using recursively applied and projected MUSIC with flexible extent estimation. Frontiers in Neuroscience, 17, 1170862. https://doi.org/10.3389/fnins.2023.1170862
Wilson, M., Hecker, L., Joos, E., Aertsen, A., Tebartz Van Elst, L., & Kornmeier, J. (2023). Spontaneous Necker-cube reversals may not be that spontaneous. Frontiers in Human Neuroscience, 17, 1179081. https://doi.org/10.3389/fnhum.2023.1179081
Hecker, L., Rupprecht, R., Tebartz Van Elst, L., & Kornmeier, J. (2021). ConvDip: A Convolutional Neural Network for Better EEG Source Imaging. Frontiers in Neuroscience, 15, 569918. https://doi.org/10.3389/fnins.2021.569918
Qureshi, M. N. I., Oh, J., & Lee, B. (2019). 3D-CNN based discrimination of schizophrenia using resting-state fMRI. Artificial Intelligence in Medicine, 98, 10–17. https://doi.org/10.1016/j.artmed.2019.06.003
Milletari, F., Navab, N., & Ahmadi, S.-A. (2016). V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation. 2016 Fourth International Conference on 3D Vision (3DV), 565–571. https://doi.org/10.1109/3DV.2016.79
Rad, N. M., & Furlanello, C. (2016). Applying Deep Learning to Stereotypical Motor Movement Detection in Autism Spectrum Disorders. 2016 IEEE 16th International Conference on Data Mining Workshops (ICDMW), 1235–1242. https://doi.org/10.1109/ICDMW.2016.0178